在分子动力学、生物医学研究和材料科学领域,Visual Molecular Dynamics(VMD)作为一款专业的分子可视化工具,凭借其强大的功能与跨平台特性,已成为科研人员不可或缺的助手。本文将系统解析VMD的软件特点、多平台下载安装流程、安全使用规范,并结合用户实际案例与未来版本前瞻,为不同需求的读者提供从入门到进阶的全方位指引。
一、软件核心功能与版本特性
作为由伊利诺伊大学研发的开源工具,VMD 1.9.4版本支持Windows/Linux/macOS系统,其核心功能包括:
- 多尺度分子可视化:支持从简单有机分子到百万级原子复杂体系的3D渲染,提供CPK、Licorice等12种显示模式(基于网页5的分子模型操作案例)
- 动态轨迹分析:可加载AMBER、GROMACS等主流分子动力学软件的轨迹文件,实现氢键追踪、能量分布计算等高级功能
- 跨平台协作能力:与NAMD、CHARMM等模拟工具无缝对接,支持远程服务器数据实时可视化
二、多平台下载与安装详解
1. 官方资源获取路径
通过VMD官网进行下载时需注意:
- 点击"Download VMD"进入版本选择页,推荐优先选择Stable Release版本
- 注册需填写机构邮箱,教育用户可申请学术授权(参考网页1的注册流程)
- Linux用户需根据CUDA版本选择对应安装包,如CUDA10对应文件名含"LINUXAMD64-CUDA10"字段
2. 分系统安装指南
Windows系统
- 双击安装程序后默认路径为C:Program FilesVMD
- 勾选Add VMD to system PATH实现全局调用
- 首次运行需通过
vmd -dispdev text命令验证OpenGL驱动兼容性
Linux系统
解压安装包(网页1/4/5综合步骤)
tar zxvf vmd-1.9.4a57.bin.LINUXAMD64-CUDA10.tar.gz
cd vmd-1.9.4a57
/configure LINUXAMD64 指定系统架构
cd src
sudo make install 需root权限写入/usr/local路径
环境变量配置(针对非默认路径)
echo 'export PATH=$PATH:/opt/vmd/bin' >> ~/.bashrc
source ~/.bashrc三、安全使用规范与常见问题
- 下载安全验证:通过
md5sum vmd.tar.gz校验文件完整性,对比官网公布的哈希值 - 权限管理建议:Linux安装时避免使用
chmod 777,推荐通过用户组权限控制 - 图形界面异常处理:若启动报错GLXBadDrawable,可尝试添加
-nogui参数启动文本模式
四、用户实际应用案例
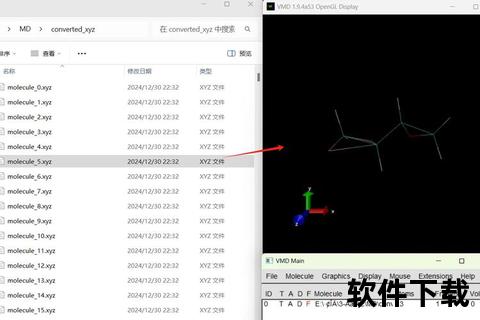
某研究团队运用VMD完成新冠病毒刺突蛋白的可视化项目时:
- 通过
mol new spike.pdb加载PDB结构文件 - 使用Representations面板设置糖基化位点的Surface渲染模式
- 利用Trajectory功能分析分子动力学模拟中的构象变化
用户反馈显示,VMD处理含600万原子的COVID-19刺突蛋白模型时,配合CUDA加速可实现实时旋转缩放操作,相比同类软件性能提升显著。
五、发展前瞻与VMD 2.0革新
2025年发布的VMD 2.0 alpha版本带来三大突破:
| 功能模块 | 技术升级 | 性能指标 |
|---|---|---|
| 界面交互 | 集成式属性面板与触摸屏优化 | 操作步骤减少40% |
| 渲染引擎 | 实时光线追踪与GLYCAM糖基可视化 | 糖蛋白渲染速度提升10⁶倍 |
| 计算架构 | 多GPU并行支持与统一内存管理 | 支持亿级原子系统 |
当前版本用户可通过GitLab参与测试计划,但需注意2.0暂未提供Windows安装包,且要求NVIDIA RTX 30系以上显卡。
六、资源获取与技术支持
推荐通过以下渠道获取权威资源:
- 官方文档库:包含300+页的VMD User's Guide与视频教程
- 开发者社区:GitHub的vmd-dev小组提供错误排查指导(参考网页3的协作开发模式)
- 学术支持计划:伊利诺伊大学提供每年两次的Workshop培训
通过本文的系统梳理,读者可全面掌握VMD的核心价值与操作要点。建议科研团队关注2.0版本的开发动态,其革新的交互设计与计算架构将重塑分子可视化领域的技术边界。